La nuova pubblicazione su Scientific Reports dei Ricercatori MAPS
Dal 15.04.2021 al 15.05.2021
È stato recentemente pubblicato, sulla rivista open access Scientific Reports di proprietà del gruppo editoriale Nature, uno studio finalizzato dai ricercatori dell’area BIO/11 del Dipartimento MAPS: il Prof. Cristian Taccioli e la dottoranda Chiara Vischioni.
La pubblicazione Successful extraction of insect DNA from recent copal inclusion: limits and perspective, è nata dalla collaborazione tra i nostri ricercatori ed il Laboratorio di estrazione di DNA antico del Professor David Caramelli del Dipartimento di Biologia dell’Università di Firenze. Altri importanti partners sono stati il Dipartimento di Genetica, Sviluppo ed Evoluzione della UCL (University College London), ed il panorama della libera professione della ricerca Patavina. L’obiettivo principale è stata l’analisi genomica di due insetti inglobati in ambra recente così da poter identificare tracce di DNA endogeno antico (aDNA) e le relative informazioni filogenetiche.
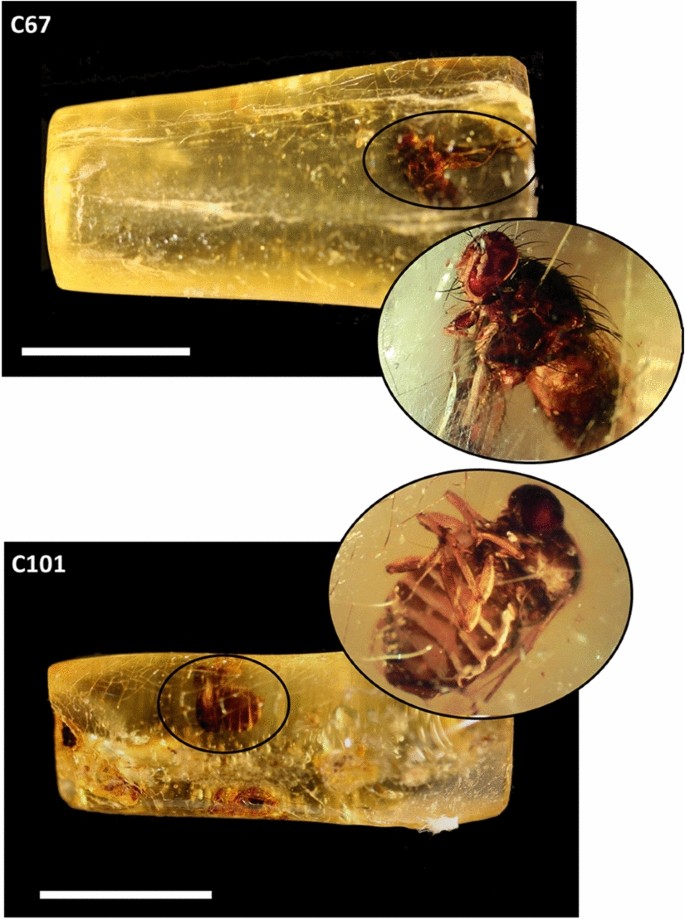
I campioni, denominati C67 e C101, entrambi di origine colombiana, sono stati analizzati e studiati attraverso diversi metodi e procedure tra cui il sequenziamento di nuova generazione (NGS) e la datazione radio carbonio. La ricerca sul DNA antico, infatti, continua ad aggiornarsi, portando gli scienziati a decidere di utilizzare costantemente nuovi approcci. Le prove, condotte dai gruppi di lavoro dei diversi Dipartimenti coinvolti, hanno consentito di giungere ad una conclusione rilevante: è possibile estrarre DNA antico da organismi inglobati in ambra recente (copale) con il limite del tempo che agisce sul genoma comportandone l’usura e la conseguente degradazione.
I campioni utilizzati, C67 e C101.
Nell’esecuzione degli esperimenti si è mostrato necessario dapprima datare i campioni, così da conoscere effettivamente l’origine temporale relativa alle misure dedotte dalle analisi radio carbonio e la successiva rimozione di contaminanti ambientali che possono degradare il DNA antico che si presenta sempre in quantità minime. Queste metodiche sono molto complicate ed, infatti, da uno dei campioni non è stato possibile estrarre DNA in quantità sufficiente per le successive analisi bioinformatiche. L’estrazione di frammenti nucleotidicidell’insetto che ha mostrato una qualità migliore a livello di conservazione, ha consentito, comunque, di creare librerie genomiche (che sono collezioni di segmenti di DNA) da utilizzare per la successiva analisi bioinformatica. I risultati di queste metodologie, a cavallo tra la biologia molecolare e l’informatica, hanno permesso di identificare la specie dell’insetto inglobato e la relativa posizione evolutiva all’interno di un albero filogenetico appositamente creato.
A partire dagli anni 2000, dopo il sequenziamento del genoma umano, anche gli studi nel campo del DNA antico (aDNA) sono diventati sempre più rilevanti, tanto da rivoluzionare molti campi della ricerca scientifica come quello del sequenziamento genomico. Negli ultimi tempi, inoltre, la tecnologia NGS ha portato i ricercatori ad analizzare diversi materiali biologici attraverso nuovi protocolli sempre più aggiornati che hanno permesso il sequenziamento, per esempio, del genoma di ominidi antichi con l’Homo sapiens neanderthalensis. In conclusione, questo articolo ha mostrato come sia possibile estrarre DNA da organismi inglobati nelle resine vegetali come copali anche se rimane l’interrogativo se sia possibile fare lo stesso utilizzando ambre fossili risalente a milioni di anni fa.
Per leggere e scaricare l'articolo, basta visitare questa pagina.